Consideration on the application of exhaustive analysis of fish with environmental DNA to the National Census on River Environments
リバーフロント研究所報告 第 31 号 2020 年 9 月
自然環境グループ 研 究 員 内藤 太輔
自然環境グループ 次 長 都築 隆禎
自然環境グループ 研 究 員 蔭山 一人
主席研究員 宮本 健也
山口大学大学院 准 教 授 赤松 良久
福岡工業大学 准 教 授 乾 隆 帝
環境 DNA 分析技術の一つメタバーコーディング解析は、特定の分類群の DNA 情報を網羅的に検出するものであり、特に魚類を対象とした研究や技術は、種判別に必要な DNA データが充実していることなどを背景として、近年の発展が著しい。本研究では、このメタバーコーディング解析の河川水辺の国勢調査(魚類調査)への導入を検討するため、現行の採捕調査とあわせて環境 DNA 調査を行い、環境区分(50〜100m スケール)、調査地区(約 1 ㎞スケール)および河川全体で、検出・確認種数を比較した。4 河川 15 調査地区 48 環境区分を対象とした調査、分析では、3 つのスケールすべてにおいて、全体平均では、環境 DNA 調査による検出数が採捕調査による確認種数を上回った。また、採捕調査による確認種数のうち環境 DNA 調査で検出できた種の割合(整合率)は、環境区分では、約 6 割程度であったのに対して、調査地区、河川全体では 7 割を越えた。一方で、大型で複雑な形のワンドでは、検出数が確認数を下回ることが確認された。さらに、下層の水をサンプルした環境 DNA 分析では、表層水のみの分析結果では漏れていた底生魚類を検出できたので、併せてその結果を報告する。
キーワード: 環境 DNA、メタバーコーディング、魚類、河川、河川水辺の国勢調査
Metabarcoding is an analysis technique for exhaustive DNA sequencing of specific taxonomic groups. Significant progress has been made with research and technologies involving analysis of fish, partly owing to abundant DNA data for discriminating species. In this study, conventional capture surveys were combined with environmental DNA sequencing to consider the application of metabarcoding to fish surveys for the National Census on River Environments. The number of detected and identified species was compared across 48 environmental compartments with a scale between 50 and 100 m, 15 survey blocks with a scale around 1 km, and four entire rivers. On an overall average, the analysis at these three different scales all resulted in a greater number of species detected by environmental DNA sequencing than by capture surveys. On the environmental compartment level, nearly 60 percent of species identified in capture surveys matched species detected by environmental DNA sequencing. The proportion exceeded 70 percent on the levels of survey blocks and entire rivers. In contrast, fewer species were detected than identified in extensive and complex lentic waters. In addition, the report mentions the detection of demersal fishes from environmental DNA sequencing with water sampled from lower layers, which were missed in the analysis of surface water.
Keywords: environmental DNA, metabarcoding, fish, rivers, National Census on River Environments
1. はじめに
河川水辺の国勢調査(以下この項では「水国調査」という)は、定期的に河川環境をモニタリングする調
査として、平成 2 年より実施され、河川管理を行う上で重要な環境情報を蓄積してきた 1)。環境に配慮した
河川管理を行うためには、これらのモニタリング調査結果を活かしつつ、新技術を用いて効率的により多く
の環境情報を取得することが求められている。
水域における環境調査の新技術として、水中に存在する生物由来の DNA を分析して生息情報を得る環境DNA 分析が、簡便かつ効率的な生物調査技術として注目されている 2)。中でも、魚類の汎用プライマーMiFish3)を用いて生息する魚類を網羅的に検出する解析法(メタバーコーディング:以下「環境 DNA 調査」という)は、煩雑なデータ処理が自動化されていること、種判別に必要な DNA データが日本産の淡水魚類の9 割以上を網羅し充実していることから、特に日本の淡水魚類の調査において有効な手段の一つと考えられている 4)。これらのことから、魚類の環境 DNA 調査は、日本の河川環境のモニタリング調査である水国調査への導入が期待されている 1)。
一方で、水国調査は、全国で統一された基準で調査され、膨大なデータが蓄積されていることから、環境DNA 調査の水国調査への導入にあたっては、これまで蓄積してきたデータとの整合性、適切なデータを得るための調査方法とその留意点などの検討が必要である。
環境 DNA 調査の水国調査への導入を念頭にした先行研究 5)では、環境 DNA 調査は同一の調査地区で実施さ
れた水国調査(採捕による魚類調査)に比べて検出・確認種数が多く有効な調査手法であることが示された一方で、環境区分(瀬・淵・ワンド等)ごとの検出・確認種の構成が両手法で異なる場合があること、水国調査で確認された魚類(特に底生魚類)が環境 DNA 調査で確認されない場合があることなどが示された。
そこで、本研究では、水国調査の魚類調査に合わせて、魚類の環境 DNA 調査を行い、先行研究 5)で課題とされた、環境 DNA 調査の結果に反映される生物情報の範囲(魚類調査の確認種との整合性が高い範囲)について、環境区分、調査地区、河川全体で比較するとともに、低層採水による底生魚類の検出について確認した。
2. 調査方法
2-1 調査対象河川
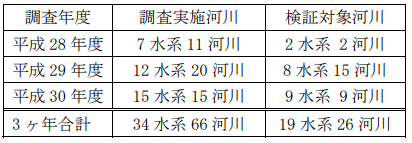
本研究では淡水区間を主な対象としたため、魚類調査が上流域に限定された、あるいは汽水域の区間が短い河川の中から、近年、河川水辺の国勢調査(以下「魚類調査」という)を実施した河川の中から、鵡川、木津川(以上、平成 29 年度)、石狩川および木曽川(以上、平成 30 年度)を対象河川とした(表-1)。なお、鵡
川および木津川は既往調査結果 5)を用いた。
表-1 調査対象河川

図-1 調査対象河川の位置図
2-2 調査の概要
環境 DNA 分析に供する検体は、各河川の国土交通大臣直轄管理区間に設定されている魚類調査の調査地区
から採水した。
採水は、魚類調査の作業前(直前から 1 週間程度前)に実施した。いずれの河川の調査においても、採水作業と魚類調査の間に大きな出水はみられなかった。
調査地区のさまざまな環境に生息する魚類を把握するために、魚類調査および採水は、調査地区内の主要な環境区分である、早瀬、平瀬、淵、ワンド・たまり、支川等で行った。
魚類調査は、平成 28 年度版河川水辺の国勢調査基本調査マニュアル[河川版](魚類調査編)6)に従い、投網、タモ網、サデ網、刺し網、定置網、地曳網、セルビン、はえ縄、電撃捕獲器、および潜水目視など、環境区分や魚類の特性に応じた調査方法により実施した。環境 DNA 調査に供する検体の採水については、各環境区分の表層水を 1L 採水した。また、底生魚類を効果的に確認する方法を検討するために、石狩川および木曽川は、淵において、表層および底層で採水を行い、結果を比較した。
2-3 環境 DNA 分析
採水検体は、塩化ベンザルコニウム 10w/v%水溶液を0.1%になるように添加、クーラーブランク(純水、1L)とともに遮光・冷蔵状態で実験室に運搬した。
以後のろ過・分析については、基本的に環境 DNA 調査・実験マニュアル ver2.17)に準拠した。
検体をろ過したフィルター(ワットマン GF/F フィルター)から DNeasy Blood & Tissue Kit (Qiagen、
Hilden、 Germany) を用いて DNA を抽出した。抽出した DNA を魚類のユニバーサルプライマーMiFish で増幅し、次世代シーケンサー (MiSeq、 Illmina) により塩基配列を決定した。塩基配列データは、MiFish パイプ
ライン 8)により解析した。得られた解析結果(配列の相同性 97%まで確認)は、公開されているデータベース
(DNA Data Bank of Japan) 9)に照合して種を確定した。なお、100 リード以下のデータは、潜在的なコンタミネーション(分析サンプル以外の DNA の混入)や不十分な反応を含むものとして、以後の解析から除外した。さらに、淡水域で検出された海産魚については、コンタミネーションや生活排水等の人間活動の影響と判断
し、データから除外した。
種の同定にあたっては、”MiFish による種の識別に注意を要する淡水魚類リスト 10)”を参照し、種判別が困難な種は、属までの同定(例:ヨシノボリ属)とした。種名は、基本的に河川水辺の国勢調査のための生物リスト(令和元年度版)11)に従った。
2-4 魚類調査と環境 DNA 調査の結果の比較
環境 DNA 分析に基づく魚類リストは、魚類調査の結果と比較し、検出力(検出種数、検出率)および精度
(魚類調査の結果との整合性)等を評価した。
環境 DNA 調査と魚類調査の結果は、以下の考え方および方法で比較した。
(1) 環境 DNA 調査の結果が反映する生物情報の範囲の検討
・環境区分ごとの確認状況:環境 DNA 調査により、環境区分(50~100m 程度の範囲)の魚類の生息状況を把できるかを検証するために、同一調査地区内の同一環境区分での結果を比較した。
・調査地区ごとの確認状況:環境 DNA 調査により、調査地区の範囲(約 1km 程度区間)の魚類の生息状況
を把握できるかを検証した。
・河川全体での確認状況:環境 DNA 調査により、河川全体の魚類生息状況を把握できるかを検証した。
(2) 底生魚類を効率的に検出する方法の検討
石狩川と木曽川を対象に、底生魚類が生息している淵において底層の河川水を採水し、表層での結果と比較した。
(3) 魚類調査と環境 DNA 調査の結果の比較方法
魚類調査と環境 DNA 調査の結果の比較では、環境 DNA調査で検出された種数(検出種数)、魚類調査で確認された種数(確認種数)、および両手法で共通して検出・確認された種数を集計した。
これらの集計から、環境 DNA 調査による検出種数を両手法で検出・確認された総種数で除した検出率、同様に魚類調査による確認種数を総種数で除した確認率、および両手法での共通検出・確認種数を魚類調査の確認種数で除した整合率 12)(式 1)を求めて評価した。
検出率、確認率は、両手法で検出・確認できた総種数を生息魚種数の真値と仮定した場合に、それぞれの手法がどの程度、検出、あるいは確認できるかを表す指標となる。
整合率は、魚類調査による確認種が環境 DNA 調査によって、どの程度検出できるのかを示す指標であり、整合率が高ければ魚類調査に対する環境 DNA 調査の代替性が高いと考えられる。

(式1)
3. 結果と考察
3-1 環境 DNA 調査および魚類調査で確認された種
石狩川と木曽川における環境 DNA 調査および魚類調査の結果(種のリスト)は既報の論文 13)に示しており、本稿では紙面上割愛した。なお、鵡川と木津川の結果は、既往調査結果 5)を用いた。
3-2 調査地区内の環境区分ごとの比較
(1) 検出種数と確認種数
各河川に設定された調査地区内の環境区分(瀬、淵、ワンド・たまり等)ごとの検出・確認種数について、魚類調査と環境 DNA 調査の結果を比較したものを図-2に示す。
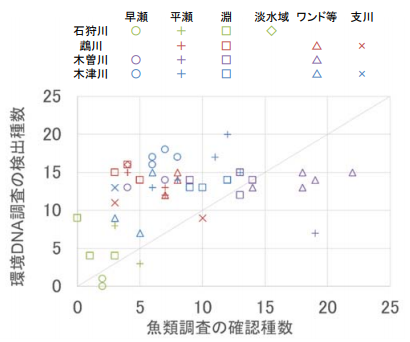
図-2 魚類調査と環境 DNA 調査の確認種数の比較
(環境区分スケール)
全 48 件中 36 件(75%)で環境 DNA 調査の検出種数が魚類調査の確認種数よりも多く、1 件で同数、11 件で少なかった。また、4 河川全ての環境区分の平均値でみると、検出種数 12.3 種で、確認種 8.0 種の約 1.5
倍であった。(図-3)
河川別には、木曽川を除く 3 河川では、環境 DNA 調査の検出数が魚類調査の確認種数を上回る件数の方が多く、特に木津川では 16 件全てで、鵡川でも調査地区3 の支川 1 件を除く 11 件で、環境 DNA 調査の方が多か
った。また、河川ごとの平均値によって検出種数と確認種数を比較しても、石狩川、鵡川、木曽川の 3 河川
はいずれも検出種数の方が多い結果となった。(図-3)
これらの結果は、環境区分の範囲(50~100m 程度)に対して、環境 DNA 調査の範囲が広いことに起因している可能性がある 4,14,15)。河川における環境 DNA 調査の検出範囲は、水理条件などによって異なることが想定されるが、いくつかの研究では上流 1 ㎞より広いと推定する結果が得られている 14,15)。
一方、木曽川では、環境 DNA 調査の検出種数の方が多かったのは、12 件中 4 件にとどまり、同数 1 件、魚類調査の確認種数より少ない環境区分が 7 件となった。また、検出種数と確認種数の平均値の比較でも検出種
数の方が少なかった。(図-3)検出種数が確認種数を下回った 7 件の内訳をみると、5 件がワンドであった。調査対象となった木曽川のワンドは、いずれも他の河川のものよりも大型(100~数百 m)で、複雑な形状のものが多かった。止水域で水の循環に乏しいワンドやたまり内では、環境 DNA の分布が偏在しており、1 箇
所の採水だけでは、生息する魚類の情報を捕捉できなかった可能性が考えられる。
図-3 環境区分ごとの確認率、検出率、整合率
(上から石狩川、鵡川、木曽川、木津川、河川別平均)
※ワンド等:ワンド・たまり
※4河川全体平均、河川別グラフ内の数字は確認種数と検出数の平均値
(2) 検出率と確認率
環境区分ごとの検出率、確認率を図-3に示す。4 河川全体の平均値では、検出率が 79.3±19.9%(平均値
±標準偏差)と確認率の 50.2±23.7%を約 30%上回り、標準偏差は小さかった。河川ごとの平均値で比較した
場合にも、木津川でほぼ同じ、他の 3 河川では検出率が確認率を大幅(22~46%)に上回った。また、標準偏
差は 4 河川全てで検出率の方が小さくなった。
木曽川については、調査地区 2 で、3 つの環境区分すべてにおいて、検出率が確認率を大きく下回った。この理由として、調査地区 2 には、網状流路が形成され、採水地点より上流にも流路の分岐があったため、一部の魚種の環境 DNA が採水から漏れた可能性があることや、確認個体数が少ない魚種の環境 DNA が検出できなかったことなどが考えられる。
(3) 整合率
環境区分ごとの整合率を図-3に示す。なお、石狩川の調査地区 1、2 の淵では、それぞれ 9 種の魚類が環境 DNA 調査で検出されたが、いずれも魚類調査の確認種数が 0 であったことから、整合率は算出できなかっ
た。
整合率の平均値は、4 河川全体では 63.3±23.4%(0~100%)、石狩川で 47.2±36.5%(0~100%)、鵡川で56.0±15.1%(29~75%)、木曽川で 57.1±18.9%(32~100%)、木津川で 79.4±14.3%(60~100%)であった。整合率の低かった石狩川は、他河川と比較して検出種数、確認種数が少ないことから、整合率のばらつきが大きくなったこと、魚類調査での確認種数が 0 となり、環境 DNA 調査の検出があったにもかかわらず、整合率が算出できなかった環境区分が 2 件あったことによる影響が考えられた。一方、木津川では、すべての環境区分において魚類調査での確認種の 6 割以上が環境 DNA 調査により検出できた。
整合率が 0%となったのは、石狩川の平瀬 1 件、早瀬 1 件の計 2 件で、平瀬では検出種数 3、確認種数 5、早瀬では検出種数 0、確認種数 2 であった。非検出となったのは、検出率が低いと報告されている 5)底生魚類であるフクドジョウ、ハナカジカ、カワヤツメ属のほか、ウグイ、ウグイ属であった。このうち、ウグイは確認個体数が 1 で、ウグイ属は幼魚の群れであったことから、環境 DNA 量が少なかったことが非検出の理由の一つとして考えられた。
一方、整合率が 100%で、確認種がすべて環境 DNA 調査で検出できたのは、石狩川 1 件、木曽川 1 件、木津川 4 件の計 6 件(全体の 13%)であったが、該当する環境区分に一定の傾向はみられなかった。
(4) まとめ
環境区分スケール(50~100m 程度の区間)の魚類調査の結果と、環境 DNA 調査の結果との比較では、4河川の平均でみると検出率が確認率を大きく上回り、ばらつきも小さい結果となった。ただし、これらの結果は検出範囲が環境区分スケールよりも広範であることに起因している可能性がある。
一方で、大型で複雑な形のワンドについては、逆に確認率が検出率を上回る結果となった。この理由として、水の流れがなく混合されにくいワンド内では、環境 DNA の分布が偏在していることが考えられた。こうした大型のワンド、あるいはたまりには、魚種数も多いことから、環境 DNA 調査では採水箇所を複数にするなどの対応が必要であろう。また、網状流路があり、流路が分岐する範囲においては、瀬や淵についても同様の対応を検討する必要があると考えられる。
また、整合率の 4 河川平均は 63.3%となり、魚類調査の確認種の 4 割程度を検出できなかった。
このように、対象範囲が大きく異なる可能性があること、整合率が 6 割程度であることから、環境区分スケールの調査は、少なくとも 1 カ所採水の環境 DNA 調査で代替することは難しいと考えられる。
3-3 調査地区ごとの比較
(1) 検出種数と確認種数
4 河川 15 調査地区における調査手法ごとの確認種数を図-4に示す。
調査地区のスケールでは、15 件中 12 件(80%)で、環境 DNA 調査の検出種数が魚類調査の確認種数よりも多く、1 件で同数、2 件で少なかった。また、4 河川全ての調査区分の平均値でみると、検出種数 18.1 種で、確認種 13.7 種の約 1.3 倍であった。
環境 DNA 調査の確認種数が魚類調査を下回った 2 件は、いずれも木曽川(調査地区 1、2)であった。これは、木曽川での環境 DNA 調査では、とくにワンド・たまりにおける検出数が魚類調査の確認種数に比べて少なかったためであると考えられる。
(2) 検出率と確認率
4 河川全体の平均値では、検出率が 85.6±11.4%(平均値±標準偏差)と確認率の 58.8±20.4%を約 30%上回り、標準偏差は小さかった。河川ごとの平均値で比較した場合、木津川では検出率が確認率を 12%下回っ
たものの、他の 3 河川ではいずれも検出率が確認率を大幅(33~50%)に上回った。また、標準偏差は木曽川
を除く 3 河川で検出率の方が小さくなった。(図-5)

図-4 調査地区ごとに集計した調査手法別の確認種数
図-5 調査地区ごとの確認率、検出率、整合率
(3) 整合率
整合率でみると(図-5)、4 河川では、調査地区の平均値は 75.6±16.2%(54~100%)であった。河川別では、石狩川および木曽川において、それぞれ 2 つの調査地区で 6 割以下となる低い整合率となった。ただ
し、石狩川については、母数となる魚類調査の確認種数が他河川と比較して少なく、魚類調査の確認種のうち、環境 DNA 調査で検出できなかった種は、該当調査地区 1、3 いずれにおいても 2 種のみであった。一方で木曽川では、該当調査地区 1、2 において、魚類調査の確認種のうち、環境 DNA 調査で検出できなかった種は、それぞれ 12 種、13 種であり、他の調査地区と比較しても多かった。これら 4 調査地区を除く 11 調査地区では、魚類調査の確認種の 7 割程度を環境 DNA 調査で検出できた。
(4) まとめ
調査地区スケールについては、環境区分スケールと同様に 4 河川の平均でみると検出率が確認率を大きく上回り、ばらつきも小さい結果となった。また、検出率、確認率とも環境区分スケールのものより高くなり、ばらつきも小さくなった。環境区分スケールに対して、より検出範囲と近いと推定される調査区分スケールにおいても検出率が確認率より 30%近く高くなったことから、対象範囲の差の影響は否定できないものの、調査区分スケールにおいて環境 DNA 調査の方がより多くの魚種を検出できる可能性が示された。
一方で河川別では、すべての調査地区に大型ワンドの環境区分がある木曽川で、検出率が確認率を下回った。
3-4 河川ごとの比較
河川単位で集計した調査手法別の確認種数を図-6に、確認率、検出率、整合率を図-7に示す。
検出・確認種数では、石狩川、鵡川、木津川で検出種数の方が多く、木曽川では確認種数の方が多くなった。
今回の調査結果では、魚類調査と環境 DNA 調査を併用した場合、魚類調査単独で行うよりも、6~13 種(13~47%)確認種数が増加した。
河川単位での検出種数、確認種数をもとにした分析では、4 河川平均で検出率 83.6%、確認率 66.1%と、環境区分、調査地区スケールと同様に検出率の方が大きくなったが、その差は 17%程度と小さくなった。また、検出率、確認率とも調査地区スケールよりも大きくなった。
河川別にみた場合には、調査地区スケールと同様に木曽川でのみ検出率が確認率を下回った。また、確認率は 4 河川全てで調査地区スケールのものより増加しているのに対して、検出率は木曽川以外の 3 河川では減少した。この理由のひとつとして、確認種に対する検出種の調査地区間での重複の多さが考えられた。
整合率については 74.3%となり、調査地区ごとの 4河川平均と大きな違いは見られなかった。ただし、河川別にみると木曽川でのみ調査地区スケールと比較して整合率が上がり、他の 3 河川では逆に下がっている。
3-5 採水層の影響
石狩川および木曽川の淵において、表層に加えて底層で採水を行い、表層の結果と比較した(図-8)。
石狩川では、調査地区 2 以外では底層の検体の方が検出種数は多かった。調査地区 2 の底層で反応が得られなかった理由は濁りによる PCR 反応の阻害等が考えられるが、詳細は不明である。
木曽川では、調査地区 4 を除き底層と表層の検体の分析から得られた種数はほとんど同じであったが、底層から採取した検体からは、同じ箇所の表層からは検出されなかったシマドジョウ属、ナマズおよびチチブ属等の底生魚類が検出された。
以上から底層での採水は、種数には大きく差がでないものの、表層だけの採水では漏れてしまう底生魚類を検出できる可能性があることを示すことができた。
4. おわりに
今回の 4 河川での調査、分析結果では、環境区分ごとの比較の場合、整合率が 6 割程度であったこと、既往の知見から環境 DNA 調査の検出範囲と環境区分のスケールが大きく異なる可能性があることから、環境区分スケールの調査は、少なくとも 1 箇所採水の環境 DNA調査で代替することは難しいと考えられる。
検出範囲と調査範囲が比較的近いと推定される調査地区スケールにおける魚種数の検出・確認では、種数の多さ、ばらつきの少なさから環境 DNA 調査の方が総じて優位であった。ただし、木曽川のように魚類調査の方が優位となるケースもあり、この要因の一つとして、大型のワンド環境があることが示された。また、網状流路があり、流路が分岐するような調査地区では、瀬、淵でも魚類調査の確認数が多くなるケースが確認
できた。
環境 DNA 調査は、様々な環境区分で採水した上で、調査地区スケール、河川単位で評価することで、魚類調査の確認種の 7 割以上をカバーする高い整合性を示し、より多くの魚種の生息を把握できる可能性が示された。
特に調査地区スケール以上で、生息する魚種を把握したい場合には、魚類調査と併用することで、より漏れの少ない高精度の調査結果を得ることが期待できる。例えば、過去の魚類調査で確認された重要種や外来種が再確認できないケースなどで、有用であろう。
また、効果的に検出率をあげる対策として、検出数が確認数より少ない傾向にある大型のワンド等では、複数個所で採水を行うことが有効と考えられた。さらに淵では、表層に加えて底層の水を分析することで、底生魚類を補完できる可能性があることを示した。
本研究では、整合率等を用いて、主に魚類調査を代替する手法として、環境 DNA 調査を魚類調査と比較したが、分析結果からは、完全な代替はできないものの相互に補完できる関係にあったことから、今後は最適な併用方法を検討することも必要と考えられる。
当面は、現行の魚類調査と合わせて環境 DNA 調査をより多くの河川、環境区分で実施し、最適な採水個所設定も含めて、検出・確認数を増やし、整合率を高くするための手法検討が必要であろう。そのうえで調査結果の利用用途、過去の調査結果との比較の可否、労力・コストなどの観点から、調査箇所の設定、あるいは同一調査日での併用や 5 年ごとの交互での実施など運用面についても検討していくことになるだろう。
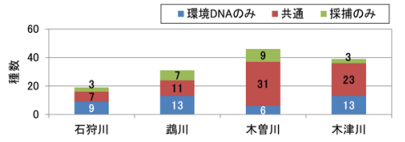
図-6 河川ごとに集計した調査手法別の確認種数
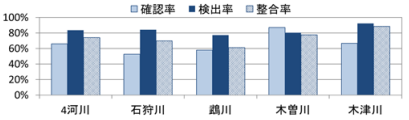
図-7 河川ごとの確認率、検出率、整合率
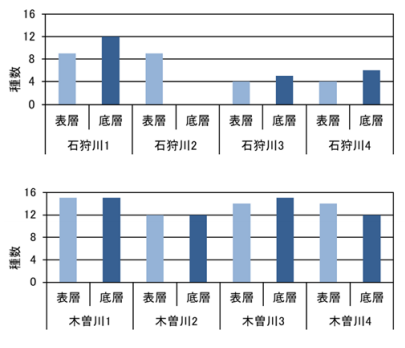
図-8 淵における表層と底層での確認種数
(上段:石狩川,下段:木曽川)
謝辞:環境 DNA 調査の実施および魚類調査結果の使用にあたり、国土交通省北海道開発局室蘭開発建設部、旭川開発建設部、近畿地方整備局木津川上流河川事務所、および中部地方整備局木曽川上流事務所に配慮をいただいた。ここに厚く御礼を申し上げる。
<参考文献>
1) 河川環境データベース:国土交通省 http://mizukoku.nilim.go.jp/ksnkankyo
2) 源利文:種特異的な環境 DNA 検出によるマクロ生物の生態調査,水環境学会誌 Vol.41(A),2018
3) Miya, Sato, Fukunaga, Poulsen, Sato, Minamoto,Yamamoto, Yamanaka, Araki, Kondoh, Iwasaki:
MiFish, a set of universal PCR primers for metabarcoding environmental DNA from fishes:detection of more than 230 subtropical marine species. Royal Society Open Science, 2:150088,2016
4) Nakagawa, H., Yamamoto, S., Sato, Y., Sado, T.,Minamoto, T., Miya, M.: Comparing local- and
regional-scale estimations of the diversity of stream fish using eDNA metabarcoding and conventional observation methods. Freshwater Biology 63,2018
5) 赤松良久,都築隆禎,横山良太,舟橋弥生,太田宗宏,畔上雅樹,内藤太輔,乾隆帝:河川水辺の国勢
調査による魚類相調査と環境 DNA メタバーコーディング解析の比較検討,土木学会論文集 B1(水工学)
Vol.74,2018
6) 河川環境データベース:平成 28 年度版河川水辺の国勢調査基本調査マニュアル[河川版](魚類調査
編),国土交通省 http://mizukoku.nilim.go.jp/ksnkankyo/mizukokuweb/system/manual.htm
7) 環境 DNA学会:環境 DNA調査・実験マニュアル ver2.1,2019
http://ednasociety.org/eDNA_manual_ver2_1_3.pdf.
8) Sato, Miya, Fukunaga, Sado and Iwasaki:MitoFish and MiFish pipeline: a mitochondrial genome database of fish with an analysis pipeline for environmental DNA metabarcoding.Molecular Biology and Evolution, 35,2018
9) Kodama, Y., Mashima, J., Kosuge, T., Kaminuma,E., Ogasawara, O., Okubo, K., Nakamura, Y.,
Takagi, T. : DNA Data Bank of Japan: 30th anniversary. Nucleic Acids Res. 4; 46(D1),2018
10) 環境:MiFish による種の識別に注意を要する淡水魚類リスト,2020
11) 河川環境データベース:河川水辺の国勢調査のための生物リスト,国土交通省
http://mizukoku.nilim.go.jp/ksnkankyo/mizukokuweb/system/seibutsuListfile.htm
12) Fujii, Doi, Matsuoka, Nagano, Sato,Yamanaka:Environmental DNA metabarcoding for
fish community analysis in backwater lakes:A comparison of capture methods. PLoS ONE
14(1): e0210357.2019
13) 内藤太輔,赤松良久,都築隆禎,横山良太,畔上雅樹,宮本健也,乾隆帝:環境 DNA による魚類の網羅的解析の河川水辺の国勢調査への導入に関する検討,河川技術論文集 第 26 巻,2020
14) 北川哲郎,村岡敬子,山田拓也,中村圭吾:河川水辺の国勢調査(魚類)における環境 DNA メタバーコーディング解析の試行事例分析,河川技術論文集, 第 26 巻,2020
15) 山口晧平,赤松良久,乾隆帝,後藤益滋,河野誉仁:河川における環境 DNA 含有物質の動態に関する基礎的研究,土木学会論文集 B1(水工学)Vol.74,2018